 Pseudomonas aeruginosa ist ein gefährliches Bakterium, verursacht schwere im Krankenhaus erworbene Infektionen und ist eine der häufigsten Todesursachen bei Menschen mit Mukoviszidose. Weltweit untersuchen Forscher das Bakterium, um herauszufinden, was es so gefährlich macht - ein wichtiger Schritt, um Menschen zukünftig vor dieser tödlichen Infektion schützen zu können. Hier verwenden wir einen systembiologischen Ansatz, um den das Zusammenspiel zwischen dem Zentralstoffwechsel und der hohen Robustheit und Widerstandsfähigkeit des Erregers zu verstehen. Wir untersuchen dazu Laborstämme und klinische Isolate aus Harnwegsinfektionen auf der Ebene metabolischer Stoffwechselflüsse. Insbesondere sind wir daran interessiert, zu verstehen, wie der Erreger oxidativen Stress übersteht, dem er während der Infektion durch die Wirtsabwehr ausgesetzt ist. Die ausgewählten Isolate sind genetisch divers, zeigen in-vitro eine heterogene Produktion von Virulenzfaktoren und stellen daher eine hervorragende Sammlung dar, um nach konservierten Stoffwechselstrategien und Nischen-spezifischen Anpassungen in diesem wichtigen menschlichen Erreger zu suchen. Hierbei arbeiten wir eng mit den Gruppen von Dieter Jahn (Technische Universität Braunschweig) und Kathrin Riedel (Universität Greifswald) zusammen.
Pseudomonas aeruginosa ist ein gefährliches Bakterium, verursacht schwere im Krankenhaus erworbene Infektionen und ist eine der häufigsten Todesursachen bei Menschen mit Mukoviszidose. Weltweit untersuchen Forscher das Bakterium, um herauszufinden, was es so gefährlich macht - ein wichtiger Schritt, um Menschen zukünftig vor dieser tödlichen Infektion schützen zu können. Hier verwenden wir einen systembiologischen Ansatz, um den das Zusammenspiel zwischen dem Zentralstoffwechsel und der hohen Robustheit und Widerstandsfähigkeit des Erregers zu verstehen. Wir untersuchen dazu Laborstämme und klinische Isolate aus Harnwegsinfektionen auf der Ebene metabolischer Stoffwechselflüsse. Insbesondere sind wir daran interessiert, zu verstehen, wie der Erreger oxidativen Stress übersteht, dem er während der Infektion durch die Wirtsabwehr ausgesetzt ist. Die ausgewählten Isolate sind genetisch divers, zeigen in-vitro eine heterogene Produktion von Virulenzfaktoren und stellen daher eine hervorragende Sammlung dar, um nach konservierten Stoffwechselstrategien und Nischen-spezifischen Anpassungen in diesem wichtigen menschlichen Erreger zu suchen. Hierbei arbeiten wir eng mit den Gruppen von Dieter Jahn (Technische Universität Braunschweig) und Kathrin Riedel (Universität Greifswald) zusammen.
- Systembiologie verschiedener P. aeruginosa Isolate
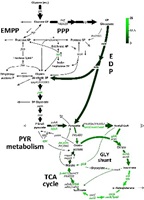
- Systemweite 13C-metabolische Fluxom-Analyse zur Erfassung der in vivo Aktivität intrazellulärer Stoffwechselwege
- Integration von Metabolom, Proteom und Transkriptom
- Aufklärung der zellulären Prozesse während Katheter-assoziierter Harnwegsinfektion
Publikationen
Dolan, S, Kohlstedt, M, Trigg, S, Ramirez, P, Kaminski, C, Wittmann, C, Welch, M (2020) Contextual flexibility in Pseudomonas aeruginosa central carbon metabolism during growth in single carbon sources. mBio 11:e02684-19. Link.
Kohlstedt, M, Wittmann, C (2019) GC-MS based 13C metabolic flux analysis resolves the cyclic glucose metabolism of Pseudomonas putida KT2440 and Pseudomonas aeruginosa PAO1. Metab. Eng. 54:35-53. Link
van Duuren, JBJH, Müsken, M, Karge, B, Tomasch, J, Wittmann, C, Häussler, S, Brönstrup, M (2017) Use of Single-Frequency impedance spectroscopy to characterize the growth dynamics of biofilm formation in Pseudomonas aeruginosa. Sci. Rep. 7:5223. PUBMED
Lassek C, Berger A, Zühlke D, Wittmann C, Riedel K (2016) Proteome and carbon flux analysis of P. aeruginosa clinical isolates from different infection sites. Proteomics. 16:1381-1385. PUBMED
Berger A, Dohnt K, Tielen P, Jahn D, Becker J, Wittmann C (2014) Robustness and plasticity of metabolic pathway flux among uropathogenic isolates of Pseudomonas aeruginosa. PLoS ONE. 9(4):e88368. PUBMED
Partner

